您現在的位置是:首頁 >動態 > 2021-04-25 06:57:59 來源:
麻省理工學院CSAIL的AI預測蛋白質的功能來自氨基酸鏈
艾未未的被挖掘,以分類 癲癇發作和預測是否乳腺癌可能轉移,但其唯一的醫療應用程序的遠。在一份計劃于5月份在國際學習代表會議上發表的學術論文中,麻省理工學院的CSAIL科學家描述了一種“計算”分解鏈狀氨基酸鏈段如何決定蛋白質功能的系統。
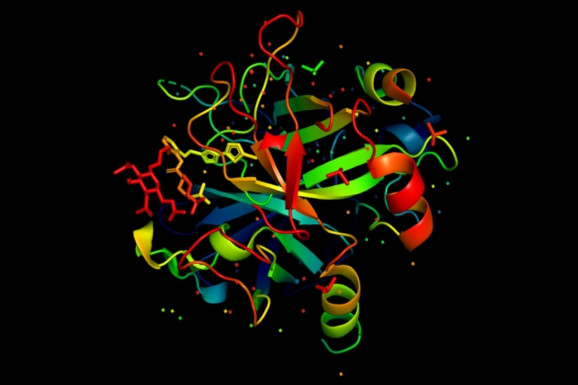
他們相信它可以用來改善蛋白質工程 - 即設計具有某些功能的新酶或蛋白質。
我希望將結構邊緣化,”CSAIL計算和生物學研究組的研究生,該論文的共同作者特里斯坦·貝普勒在一份聲明中說。“我們想知道蛋白質的作用,知道結構對此非常重要。但是,我們能否預測僅給出其氨基酸序列的蛋白質的功能?動機是遠離特異性預測結構,轉向[發現]氨基酸序列如何與功能相關。“
正如Bepler及其同事所解釋的那樣,蛋白質的行為 - 包含上述氨基酸鏈,每個都通過肽鍵緊密連接 - 很難用機器學習來預測。(也就是說,谷歌的DeepMind在12月份使用AlphaFold取得了令人矚目的成就。)只記錄了數以萬計的數百萬個三維折疊蛋白質形狀,并且氨基酸序列通常具有相似的結構,因此很難區分小說并重復結果。
因此,論文的作者采用了不同的方法:將預測的蛋白質結構直接編碼為表示。具體而言,他們在開源蛋白質結構分類(SCOP)中培訓了大約22,000種標記蛋白質的AI系統)數據庫,并為每一對計算得分,表明兩者在結構上有多接近。然后,他們提供了模型隨機對蛋白質和嵌入(即,數學表示)他們的氨基酸序列,從中學習預測他們的3D結構可能有多相似。最后,他們讓模型比較兩個相似性得分,以確定哪些成對的嵌入共享蛋白質結構,并構建它以同時預測“內容圖”,指示每個氨基酸與蛋白質結構中的其他氨基酸的距離。
所有這些工作的結果?一個端到端的系統,給定氨基酸鏈作為輸入,為蛋白質中的每個氨基酸位置產生嵌入 - 嵌入其他模型可用于預測所述氨基酸的功能。在一項實驗中,研究人員訓練了一種模型來比以前的方法更準確地預測跨膜和非跨膜片段。
“我們的模型允許我們將已知蛋白質結構的信息傳遞給結構未知的序列。使用我們的嵌入作為功能,我們可以更好地預測功能并實現更有效的數據驅動蛋白質設計,“Bepler說。“從高層次來說,這種類型的蛋白質工程是目標。因此,我們的機器學習模型使我們能夠從相對較少的已知結構中學習蛋白質折疊的“語言” - 最初的“圣杯”問題之一。